脂质纳米颗粒(LNPs)是当前mRNA疗法领域的主流递送载体,其递送效能与颗粒内部的脂质结构及相态排布密切相关,直接决定了载体的稳定性、包封效率以及mRNA释放效率。动态光散射(DLS)、zeta电位测量等常规表征技术虽能提供粒径和表面电荷等信息,却无法揭示脂质与mRNA在纳米颗粒内部的具体排列方式。而像冷冻电镜(cryo-EM)、核磁共振(NMR)这类高分辨率表征方法虽可提供上述信息,但这类技术通常通量低,并不适用于需要快速筛选大量配方条件的研发初期阶段。
小角X射线散射技术(SAXS)可有效弥补这一缺口,它可以提供脂质纳米颗粒的结构、层状特征与内部排布提供了详细见解[1-6]。FDA针对脂质体药物制剂的指导原则中,已将类似的结构参数列为关键质量属性[7]。SAXS技术还能直接解析mRNA的封装状态[4,6,8,9]、脂质双层结构[10]、物相鉴定[1,2,8,11-12],以及在不同pH[2,11,12]、温度[2]和脂质组成[1,2,5,10]条件下的结构稳定性。若结合广角X射线散射技术(WAXS),还可进一步识别制剂中的晶相结构[13],从而为配方优化与质量控制评估提供有力支撑。
近期,Sanofi的研究人员利用实验室SAXS/WAXS技术,成功表征了PEG化mRNA-LNPs,并揭示了其结构特征图谱与递送效率之间的内在关联[1]。
为探究PEG化对mRNA-脂质纳米颗粒内部结构的影响,他们选取了三类配方进行比较分析。其中两种作为参照体系:一种含DMG–PEG2000(Std1),另一种含DMG–PEG5000(Std2)。前者是LNP研发中应用广泛的标准配方,而后者则是通过数十种PEG化脂质筛选后确定的配方。第三种为优化配方(OPT),其同样采用 DMG–PEG5000,但通过实验设计(DoE)方法进行了进一步的配方改良。该策略能够系统性地探究脂质组分的影响,从而筛选出最利于 mRNA高效递送的结构特征。通过对比这三种代表性体系,研究人员可以直接获得PEG链长与组分优化如何影响层状与六方相结构的动态平衡、结构有序度,并最终影响与递送效率密切相关的各项性能[1]。
SAXS揭示mRNA-LNPs的层状与六方相结构
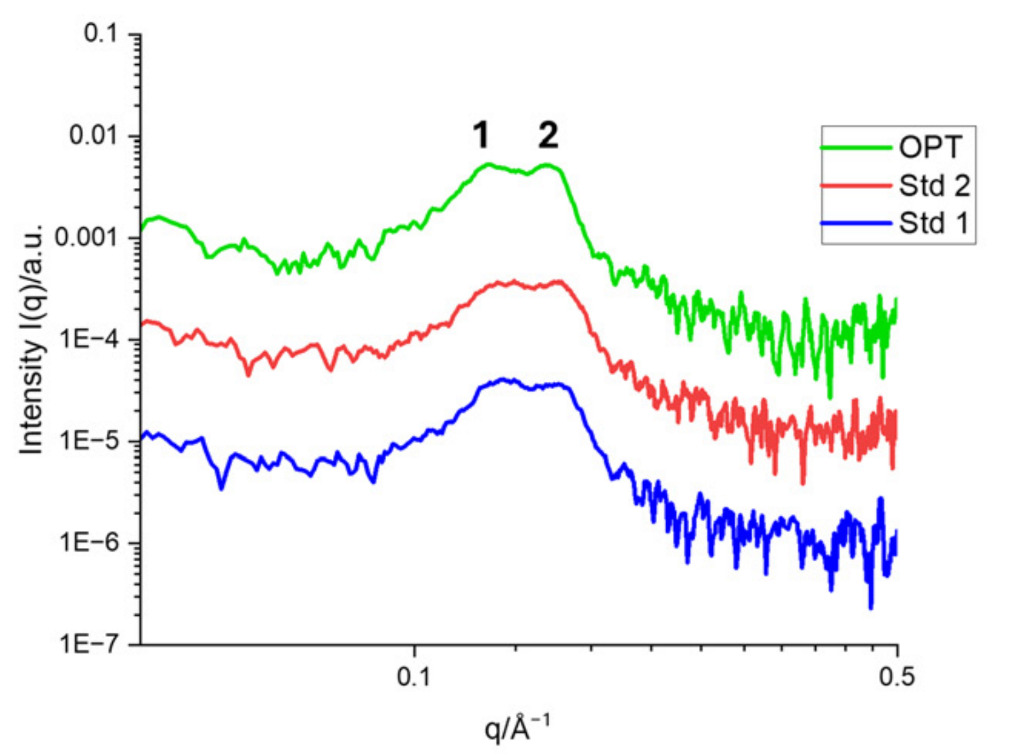
通过对三种配方的SAXS图谱分析,其中有两种显著的结构特征信号:位于q ≈ 0.13 Å⁻¹处的布拉格峰对应于反六方相结构,而q ≈ 0.16 Å⁻¹处的峰则对应于多层层状相结构(图1)。冷冻电镜结果进一步证实了这两种物相在脂质体内的共存,凸显了SAXS技术在溶液物相鉴定中的可靠性。在优化配方(OPT)中,层状相的重复间距约为4.0nm,六方相间距约为4.9nm,该数据与冷冻透射电镜的测量结果高度一致。
Figure 1. 在10%海藻糖溶液中,OPT mRNA-LNP配方与两种标准配方Std1和Std2的SAXS图谱对比。
来源: Nanoscale, 2025 DOI: 10.1039/D5NR00433K
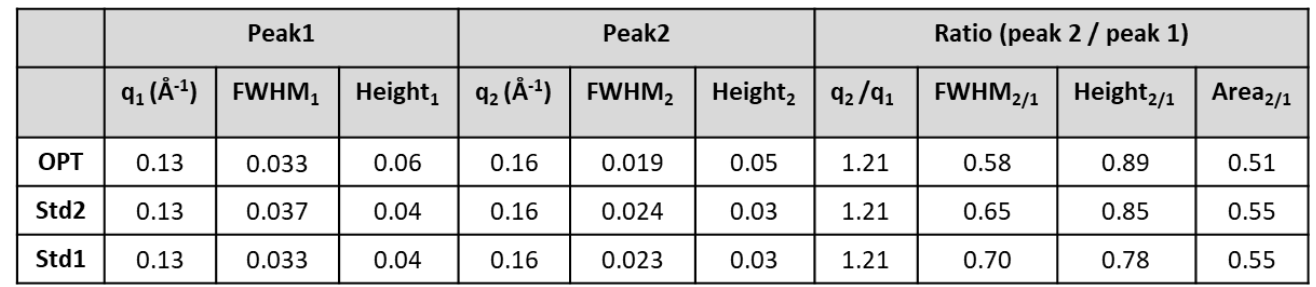
除物相鉴定外,SAXS技术还成功捕捉到纳米结构有序度的差异。与两种参照配方(Std1、Std2)相比, OPT的反六方相峰更尖锐、强度更高,半峰宽更窄(表1)。这表明存在更大且有序度更高的六方相结构。与此同时,OPT配方的层状相峰相对较弱,如较小的Area2/1所示,反映出结构明显向具有膜融合活性的六方相转变。该相态因能有效促进膜融合和内体逃逸而广为人知,二者均是实现mRNA高效释放的关键步骤。
Table 1. 在10%海藻糖溶液中,OPT、Std2及Std1配方的SAXS峰特征对比。来源: Nanoscale, 2025 DOI: 10.1039/D5NR00433K
SAXS揭示pH驱动mRNA释放的结构转变机制
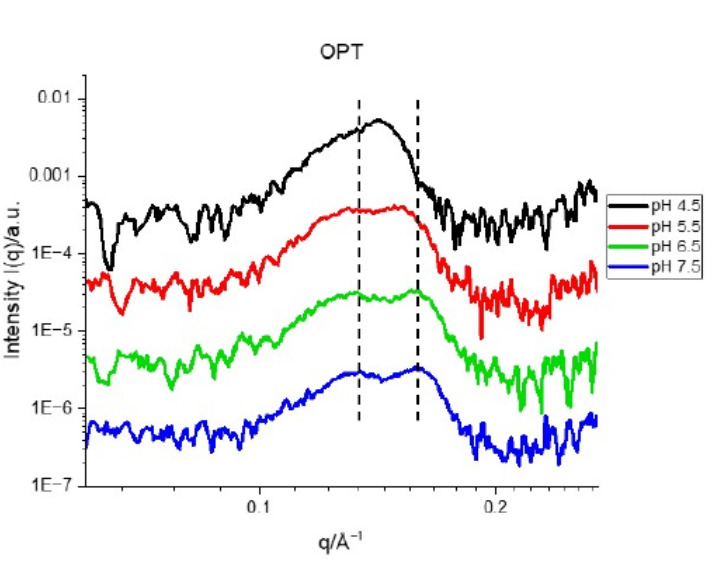
为探究pH值这一模拟内体环境的关键参数对结构的影响,研究人员采用SAXS技术监测三种配方在不同pH条件下的结构转变过程。
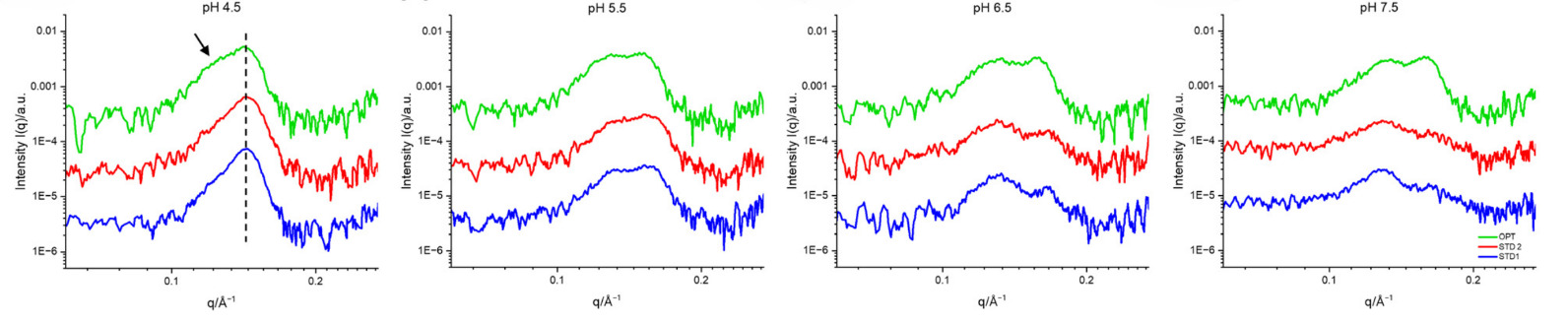
在接近生理pH(7.5)条件下,优化配方(OPT)的SAXS图谱同时呈现出显著的层状相与六方相结构特征,而两种参照配方(Std1、Std2)仅呈现较弱的多层状相信号(图2)。
随着pH值降低,Std1与Std2配方发生了显著的结构重组——其层状相与六方相衍射峰逐渐变得更为尖锐,体现出结构从相对无序向有序的转变。与此形成鲜明对比的是,OPT配方即使在pH低至4.5的条件下,依然保持着最强的反六方相衍射峰信号(图2-3)。这一结果说明OPT体系在生理条件下已预先形成了更稳定、更有序的融合活性结构,从而为其实现高效的内体逃逸与mRNA释放提供了结构基础。
Figure. 2. OPT、Std2与Std1配方在不同pH值柠檬酸-磷酸盐缓冲液中的SAXS图谱对比。来源: Nanoscale, 2025 DOI: 10.1039/D5NR00433K
Figure 3. OPT mRNA-LNP配方在不同pH值柠檬酸-磷酸盐缓冲液中的SAXS图谱变化。来源: Nanoscale, 2025 DOI: 10.1039/D5NR00433K
此外,SAXS数据还显示,在酸性pH条件下,布拉格峰位置向更低q值方向移动,对应的晶面间距随之增大(图3)。这种结构膨胀意味着纳米颗粒吸入了水分,而该结构调整也进一步体现出其对内体环境的响应特性。
SAXS/WAXS揭示从纳米结构与递送的核心结论
研究结果充分证明,实验室级SAXS/WAXS技术能够有效解析LNPs的内部组织结构,可定量获取脂质层重复间距(晶格间距)与结构规整度(纳米结构有序性)等关键参数。这些发现与冷冻电镜结果高度一致,同时兼具高通量、溶液原位表征的技术优势。
重要的是,SAXS/WAXS技术不仅能够识别出明确的结构特征图谱,还可追踪这些结构随pH变化的动态转变过程,这一特性对于理解内体逃逸过程至关重要。优化后的配方呈现出更尖锐、有序度更高的六方相结构,且结构明显偏向于具有膜融合活性的物相,这与实验中观察到的mRNA释放效率提升现象相符。
通过直接建立纳米结构与递送性能之间的联系,SAXS/WAXS技术能够支持多条件下配方的快速筛选、构效关系研究,以及工业化生产流程中的质量评估。总之,SAXS/WAXS已成为设计、优化及质控下一代mRNA-LNP疗法的可靠高通量工具。
The research was originally published in the following article: [1] Liu, L., Kim, J.H., Li, Z., Sun, M., Northen, T., Tang, J.,. Mcintosh, E., Karve, S., DeRosa, F. PEGylated lipid screening, composition optimization, and structure-activity relationship determination for lipid nanoparticle-mediated mRNA delivery. Nanoscale, 2025 17, DOI: 10.1039/d5nr00433k.
参考文献
[1] Liu, L., Kim, J.H., Li, Z., Sun, M., Northen, T., Tang, J., Mcintosh, E., Karve, S., DeRosa, F. PEGylated lipid screening, composition optimization, and structure-activity relationship determination for lipid nanoparticle-mediated mRNA delivery. Nanoscale, 2025, 17, 11329–11344, DOI: 10.1039/d5nr00433k.
[2] Pattipeiluhu, R., Zeng, Y., Hendrix, M.M.R.M., Voets, I.K., Kros, A., Sharp, T.H. Liquid crystalline inverted lipid phases encapsulating siRNA enhance lipid nanoparticle mediated transfection. Nat. Commun. 2024, 15, 1303. DOI: 10.1038/s41467-024-45666-5.
[3] Dao, H.M., AboulFotouh, K., Hussain, A.F., Marras, A.E., Johnston, K.P., Cui, Z., Williams, R.O 3rd. Characterization of mRNA lipid nanoparticles by tlectron density mapping reconstruction: X-ray scattering with density from solution scattering (DENSS) algorithm. Pharm. Res. 2024, 41, 501-512. DOI: 10.1007/s11095-024-03671-9.
[4] Gilbert, J., Sebastiani, F., Arteta, M.Y., Terry, A., Fornell, A., Russell, R., Mahmoudi, N., Nylander, T. Evolution of the structure of lipid nanoparticles for nucleic acid delivery: From in situ studies of formulation to colloidal stability. J. Colloid Interface Sci. 2024, 660, 66-76. DOI: 10.1016/j.jcis.2023.12.165.
[5] Wilhelmy, C., Keil, I.S., Uebbing, L., Schroer, M.A., Franke, D., Nawroth, T., Barz, M., Sahin, U., Haas, H., Diken, M., Langguth, P. Polysarcosine-Functionalized mRNA Lipid Nanoparticles Tailored for Immunotherapy. Pharmaceutics, 2023, 15, 2068. DOI: 10.3390/pharmaceutics15082068.
[6] US Food and Drug Administration. Liposome Drug Products: Chemistry, Manufacturing, and Controls; Human Pharmacokinetics and Bioavailability; and Labeling Documentation. In Guidance for Industry.(CDER); Food and Drug Administration: Rockville, MD, USA, 2018.
[7] Cui, L., Hunter, M.R., Sonzini, S., Pereira, S., Romanelli, S.M., Liu, K., Li, W., Liang, L., Yang, B., Mahmoudi, N., Desai, A.S. Mechanistic studies of an automated lipid nanoparticle reveal critical pharmaceutical properties associated with enhanced mRNA functional delivery in vitro and in vivo. Small, 2022, 18, e2105832. DOI: 10.1002/smll.202105832.
[8] Patel, S., Ashwanikumar, N., Robinson, E., Xia, Y., Mihai, C., Griffith, J.P 3rd, Hou, S., Esposito, A.A., Ketova, T., Welsher, K., Joyal, J.L., Almarsson, Ö., Sahay, G. Naturally-occurring cholesterol analogues in lipid nanoparticles induce polymorphic shape and enhance intracellular delivery of mRNA. Nat Commun. 2020, 11, 983. DOI: 10.1038/s41467-020-14527-2.
[9] Kulkarni, J.A., Darjuan, M.M., Mercer, J.E., Chen, S., van der Meel, R., Thewalt, J.L., Tam, Y.Y.C., Cullis, P.R. On the Formation and morphology of lipid nanoparticles containing ionizable cationic lipids and siRNA. ACS Nano. 2018, 12, 4787-4795. DOI: 10.1021/acsnano.8b01516.
[10] Hammel, M., Fan, Y., Sarode, A., Byrnes, A.E., Zang, N., Kou, P., Nagapudi, K., Leung, D., Hoogenraad, C.C., Chen, T., Yen, C.W., Hura, G.L. Correlating the structure and gene silencing activity of oligonucleotide-loaded lipid nanoparticles using small-angle X-ray scattering. ACS Nano. 2023, 17, 11454-11465. DOI: 10.1021/acsnano.3c01186.
[11] Xu, Z., Seddon, J.M., Beales, P.A., Rappolt, M., Tyler,, A.I.I. Tyler Breaking isolation to form new networks: pH-triggered changes in connectivity inside lipid nanoparticles J Am Chem Soc, 2021, 143, pp. 16556-16565. DOI: 10.1021/jacs.1c06244.
[12] Uebbing L, Ziller A, Siewert C, Schroer MA, Blanchet CE, Svergun DI, Ramishetti S, Peer D, Sahin U, Haas H, Langguth P. Investigation of pH-responsiveness inside lipid nanoparticles for parenteral mRNA application using small-angle X-ray scattering. Langmuir. 2020, 36, 13331-13341. DOI: 10.1021/acs.langmuir.0c02446.
[13] Yanez Arteta, M., Kjellman, T., Bartesaghi, S., Wallin, S., Wu, X., Kvist. A.J, Dabkowska, A., Székely, N., Radulescu, A., Bergenholtz, J., Lindfors, L. Successful reprogramming of cellular protein production through mRNA delivered by functionalized lipid nanoparticles. Proc. Natl. Acad. Sci. 2018, 115, E3351–E3360. DOI: 10.1073/pnas.1720542115.